

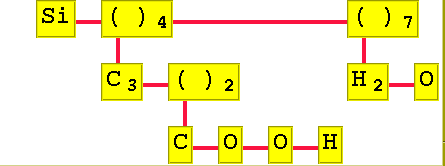
Molekyl: Si(C3(COOH)2)4(H2O)7 Molekyl:och i molekylfönstret ritar programmet ut formelstrukturen så här:
class Ruta:
atom="( )"
num=1
next=None
down=None
Funktionen readgroup skapar först en sådan tomruta med
rutan = Ruta() och anropar readatom och
readnum för att kunna sätta in rätt värden på atom
och num. Om det är en parentesgrupp ska readgroups
anrop till readmol returnera en delmolekyl som sätts under
rutan.down.
När readgroup är klar returnerar den rutan till anropet
mol=readgroup()
som görs allra först i readmol. Vad som ska göras med
mol.next får du själv tänka ut. Slutligen returnerar
readmol den färdiga strukturen till readformel
som returnerar den till huvudprogrammets anrop
mol = readformel()
där mol pekar högst upp till vänster på syntaxträdet.
show från. Sedan ska
show(mol)rita upp molekylbilden i ett eget fönster. Bilden ritas förstås rekursivt, och du ska formulera den rekursiva tanke som används. Om du inte kommer på den själv kanske det hjälper att kolla molgrafikkoden. Om programmet avslutas direkt hinner man inte se grafiken blinka förbi. Se därför till att ha en slinga för inmatning av flera formler.
Molekylvikten kan beräknas rekursivt med anropet weight(mol).
Formulera först en mycket rekursiv tanke för vikten och programmera den sedan!
Modifiera molgrafiken så att vikten skrivs ut i samma fönster.